I grafici di dispersione (scatter plot o scatterplot) forniscono un ottimo esempio delle potenzialità delle funzioni del pacchetto ggplot2 [1] nella rappresentazione grafica dei dati. Funzioni che possono risultare ostiche a chi le affronta per la prima volta, ma che consentono risultati che non si riescono a ottenere impiegando esclusivamente le funzioni base di R [2].
Oltre al pacchetto ggplot2 bisogna avere installato – o bisogna scaricare anche – il pacchetto DAAG [3] che contiene i dati impiegati nell'esempio e il pacchetto gridExtra che nel nostro caso è necessario per combinare più grafici in una sola figura.
Copiate e incollate nella Console di R questo script e premete ↵ Invio.
# SCATTERPLOT con ggplot
#
library(DAAG) # carica il pacchetto DAAG che include il set di dati ais
library(ggplot2) # carica il pacchetto per la grafica
library(gridExtra) # carica il pacchetto per combinare i grafici in una sola figura
#
# scatterplot
plot1 <- ggplot(ais, aes(x=rcc, y=hg)) + geom_point()
#
# con regressione loess
plot2 <- ggplot(ais, aes(x=rcc, y=hg)) + geom_point() + geom_smooth()
#
# con regressione lineare
plot3 <- ggplot(ais, aes(x=rcc, y=hg)) + geom_point() + geom_smooth(method="lm")
#
# con la densità di distribuzione
plot4 <- ggplot(ais, aes(x=rcc, y=hg)) + geom_point(aes(color="red")) + geom_density_2d() + theme_classic() + labs(title="Densità di distribuzione \n tutti i casi", x="Eritrociti in 10^6/μL", y="Emoglobina in g/dL") + theme(plot.title=element_text(hjust=0.5), legend.position="none")
#
# combina i grafici in una sola figura
grid.arrange(plot1, plot2, plot3, plot4, nrow=2, ncol=2)
#
Lo script è semplice: dopo avere caricato i pacchetti necessari, sono generati quattro grafici che sono salvati separatamente per essere poi, al termine, combinati in una sola figura.
Per realizzare il primo grafico (plot1) viene impiegata la funzione ggplot() che inizializza l'oggetto al quale sono collegate con il segno più [+] le successive funzioni, che sviluppano la grafica e che prevede:
→ come primo argomento il nome della tabella che contiene i dati (ais) [3];
→ come secondo argomento la funzione aes() che specifica la variabile da riportare in ascisse (rcc) e la variabile da riportare in ordinate (hg), che sono rispettivamente la concentrazione degli eritrociti nel sangue espressa in milioni per microlitro di sangue (10^6/µL) e la concentrazione di emoglobina espressa in grammi per decilitro di sangue (g/dL).
Alla funzione ggplot() viene quindi collegata con il segno più [+] la funzione geom_point() che realizza il grafico xy lasciando per tutti gli argomenti i valori previsti di default.
Da notare subito che questa è la struttura base che rimane identica in tutti e quattro i grafici realizzati, che si differenziano tra loro per le componenti grafiche via via aggiunte.
Il secondo grafico (plot2) quindi riprende tal quale la precedente struttura base e aggiunge semplicemente tramite la funzione geom_smooth(), la regressione loess prevista di default, con i suoi limiti di confidenza (la regressione loess è una regressione che collega tra loro una serie di regressioni polinomiali, quelle che localmente meglio si adattano ai dati).
Nel terzo grafico (plot3) impiegando l'argomento method la regressione loess è sostituita con la usuale retta di regressione ("lm"), riportando anche in questo caso i limiti i confidenza. Se non li volete rappresentare correggete la riga di codice sostituendola completamente con la seguente o semplicemente aggiungendo quanto qui riportato in colore
# con regressione lineare
plot3 <- ggplot(ais, aes(x=rcc, y=hg)) + geom_point() + geom_smooth(method="lm", se=FALSE)
#
Il quarto grafico (plot4) riprende la struttura base aggiungendo:
→ con aes(color="red") un colore ai punti che rappresentano i dati;
→ con la funzione geom_density_2d() la rappresentazione sul piano dei livelli di densità nella distribuzione dei dati;
→ con la funzione theme_classic() il cambiamento di stile che risulta evidente nell'ultimo grafico;
→ con la funzione labs() il titolo e le etichette degli assi, Da notare il simbolo \n che all'interno di una stringa di testo determina il ritorno a capo;
→ con la funzione theme() il codice (abbastanza arzigogolato) che determina la centratura del titolo e il valore "none" per l'argomento legend.position che determina la posizione della legenda, che pertanto non viene riportata.
Infine con la funzione grid.arrange() i quattro grafici sono combinati in un'unica figura.
# SCATTERPLOT con ggplot
# colori differenziati in base a un fattore
#
library(DAAG) # carica il pacchetto DAAG che include il set di dati ais
library(ggplot2) # carica il pacchetto per la grafica
library(gridExtra) # carica il pacchetto per combinare i grafici in una sola figura
#
# scatterplot per sesso
plot1 <- ggplot(ais, aes(x=rcc, y=hg)) + geom_point(aes(color=sex))
#
# con simboli dei punti differenziati
plot2 <- ggplot(ais, aes(x=rcc, y=hg)) + geom_point(aes(color=sex, shape=sex)) + theme(legend.position="none")
#
# con rette di regressione e limiti di confidenza
plot3 <- ggplot(ais, aes(x=rcc, y=hg)) + geom_point(aes(color=sex, shape=sex)) + theme(legend.position="none") + geom_smooth(aes(group=sex, color=sex), method="lm", se=TRUE)
#
# con rette di regressione estese
plot4 <- ggplot(ais, aes(x=rcc, y=hg)) + geom_point(aes(color=sex, shape=sex)) + theme(legend.position="none") + geom_smooth(aes(group=sex, color=sex), method="lm", se=FALSE, fullrange=TRUE)
#
# combina i grafici in una sola figura
grid.arrange(plot1, plot2, plot3, plot4, nrow=2, ncol=2)
#
Per realizzare il primo grafico (plot1) viene impiegata la struttura base già vista nello script precedente, ma con la semplice aggiunta a geom.point della funzione aes() che con l'argomento color posto uguale a sex riporta i dati con un colore differenziato in base al sesso.
Il secondo grafico (plot2) riprende tal quale il codice precedente ma:
→ alla funzione geom_point() aggiunge l'argomento shape=sex che automaticamente riporta un simbolo differente in base al sesso;
→ con la funzione theme() esclude dal grafico la legenda.
Il terzo grafico (plot3) è identico al secondo ma con la funzione geom_smooth() aggiunge la retta di regressione ordinaria (method="lm") con i limiti di confidenza (se=TRUE) separatamente (group=sex) e con un colore diverso (color=sex) per ciascun sesso.
Il quarto grafico (plot4) riprende esattamente il codice del terzo ma con se=FALSE esclude la rappresentazione dei limiti di confidenza e con fullrange=TRUE riporta la retta di regressione a tutto campo e al di la dei limiti dei dati osservati (per inciso si ricorda che, a meno di casi particolari e adeguatamente giustificati, l'estrapolazione non andrebbe mai fatta, ma viene qui riportata per completezza).
Infine con la funzione grid.arrange() i quattro grafici sono di nuovo combinati, per semplicità, in un'unica figura.
Ora copiate e incollate nella Console di R questo script e premete ↵ Invio.
# SCATTERPLOT con ggplot
# dati con densità delle distribuzioni
#
library(DAAG) # carica il pacchetto DAAG che include il set di dati ais
library(ggplot2) # carica il pacchetto per la grafica
library(gridExtra) # carica il pacchetto per combinare i grafici in una sola figura
#
# scatterplot con ellissi
plot1 <- ggplot(ais, aes(x=rcc, y=hg)) + geom_point(aes(color=sex, shape=sex)) + theme(legend.position="none") + stat_ellipse(aes(color=sex))
#
# con densità di distribuzione della y
plot2 <- ggplot(ais, aes(y=hg, fill=sex)) + geom_density(alpha=0.5) + theme(legend.position="none")
#
# con densità di distribuzione della x
plot3 <- ggplot(ais, aes(x=rcc, fill=sex)) + geom_density(alpha=0.5) + theme(legend.position="none")
#
# con livelli di densità separati per sesso
plot4 <- ggplot(ais, aes(x=rcc, y=hg)) + geom_point(aes(color=sex, shape=sex)) + theme(legend.position="none") + geom_density_2d(aes(color=sex))
#
# combina i grafici in una sola figura
grid.arrange(plot1, plot2, plot3, plot4, nrow=2, ncol=2)
#
Il primo grafico (plot1) riprende le funzioni già note, ma aggiunge, con la funzione stat_ellipse(), le ellissi che riportano i limiti di confidenza al 95% delle distribuzioni dei dati, separate per ciascun sesso e ulteriormente analizzate con il secondo e il terzo grafico.
Il secondo grafico (plot2) riporta in ordinate per la variabile emoglobina (y=hg) e separatamente per ciascun sesso (fill=sex), con la funzione geom_density(), il kernel density plot, con un colore trasparente al 50% (alpha=0.5) per consentirne la sovrapposizione. Da notare due aspetti non banali gestiti automaticamente dalle funzioni qui impiegate: gli assi sono ruotati nella posizione corretta e per l'asse delle ordinate la scala nella quale sono espressi i valori di concentrazione dell'emoglobina (hg) è allineata con quella del primo grafico.
Il terzo grafico (plot3) riporta in modo analogo ma in ascisse il kernel density plot per sesso della variabile concentrazione degli eritrociti (x=rcc). Da notare che anche in questo caso gli assi sono automaticamente ruotati nella posizione corretta e per l'asse delle ascisse la scala nella quale sono espressi i valori di concentrazione degli eritrociti (rcc) è automaticamente allineata con quella del primo grafico.
In definitiva i primi tre grafici formano un tutt'uno organico – con le variabili rappresentate contemporaneamente sia singolarmente in forma di grafico xy sia in forma di grafici che ne riportano in continuo la densità (kernel density plot) – che fornisce un'analisi molto interessante dei dati, mostrando come la distribuzione dei valori di concentrazione dell'emoglobina nel sangue differisce nei due sessi.
Il quarto grafico (plot4) riprende esattamente il primo aggiungendo questa volta al posto delle ellissi con la funzione geom_density_2d() la rappresentazione sul piano dei livelli di densità nella distribuzione dei dati.
Se vedendo il grafico notate che preferireste (io lo preferisco di gran lunga) che i due kernel density plot fossero riferiti al grafico in basso a destra, niente di più facile, copiate e incollate nella Console di R questo script e premete ↵ Invio.
# SCATTERPLOT con ggplot
# dati con densità delle distribuzioni
#
library(DAAG) # carica il pacchetto DAAG che include il set di dati ais
library(ggplot2) # carica il pacchetto per la grafica
library(gridExtra) # carica il pacchetto per combinare i grafici in una sola figura
#
# scatterplot con ellissi
plot1 <- ggplot(ais, aes(x=rcc, y=hg)) + geom_point(aes(color=sex, shape=sex)) + theme(legend.position="none") + stat_ellipse(aes(color=sex))
#
# con densità di distribuzione della y
plot2 <- ggplot(ais, aes(y=hg, fill=sex)) + geom_density(alpha=0.5) + theme(legend.position="none") + scale_x_reverse()
#
# con densità di distribuzione della x
plot3 <- ggplot(ais, aes(x=rcc, fill=sex)) + geom_density(alpha=0.5) + theme(legend.position="none")
#
# con livelli di densità separati per sesso
plot4 <- ggplot(ais, aes(x=rcc, y=hg)) + geom_point(aes(color=sex, shape=sex)) + theme(legend.position="none") + geom_density_2d(aes(color=sex))
#
# combina i grafici in una sola figura
grid.arrange(plot1, plot3, plot2, plot4, nrow=2, ncol=2)
#
Come si vede il codice è identico al precedente, a parte il fatto che dovete:
→ scambiare tra di loro i grafici (plot3, plot2,) quando li combinate in un'unica figura (ultima riga di codice), portando quindi il kernel density plot degli eritrociti (rcc) in alto a destra e quello dell'emoglobina (hg) in basso a sinistra;
→ aggiungere con (+) al plot2 la funzione scale_x_reverse() per invertire la scala dell'asse delle ascisse e quindi la rappresentazione del kernel density plot.
Ricordate, perché prima o poi vi tornerà utile, che qualora fosse necessario potete anche invertire la scala dell'asse delle ordinale con scale_y_reverse() e scambiare di posizione gli assi con coord_flip().
Concludiamo con questo script, copiatelo e incollatelo nella Console di R e premete ↵ Invio:
# GRAFICO DI DISPERSIONE con ggplot
# colori differenziati in base a un fattore
#
library(DAAG) # carica il pacchetto che include il set di dati ais
library(ggplot2) # carica il pacchetto per la grafica
#
windows() # apre e inizializza una nuova finestra grafica
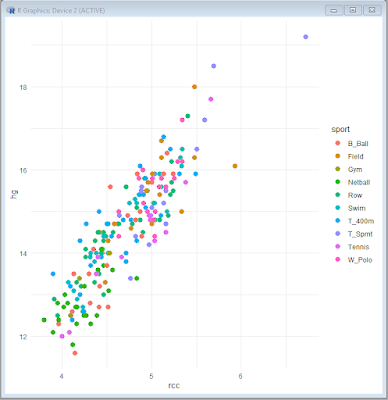
ggplot(ais, aes(x=rcc, y=hg, color=sport)) + geom_point(size=3) + theme_minimal() # traccia il grafico
#
Ed ecco il grafico
che ci consente di mostrare come la funzione ggplot() sia in grado di coniugare semplicità e potenza nella rappresentazione dei grafici di dispersione (scatterplot).
Sono infatti sufficienti come argomenti della funzione solamente:
→ la tabella che include i dati (ais);→ la x (rcc) e la y (hg) da rappresentare;
→ l'indicazione della variabile "fattore" (sport) che contiene i valori (lo sport praticato) sulla base dei quali attribuire ai punti un differente colore (color=);
mentre sono facoltativi e aggiunti solamente per completezza la dimensione dei punti (size=3) e la funzione theme_minimal() che toglie il colore grigio di sfondo.
Conclusione: questa breve presentazione ha voluto evidenziare come le funzioni incluse nel pacchetto ggplot2 possono sia essere impiegate in modo agevole, sia fornire un importante aiuto nella introspezione dei dati e nella comunicazione dell'informazione in essi contenuta [4].
----------
[1] Vedere la documentazione e il manuale di riferimento su:
https://cran.r-project.org/web/packages/ggplot2/index.html
[2] Vedere il post Grafici di dispersione (scatterplot).
[3] Vedere il post Il set di dati ais nel quale trovate anche come caricare i dati senza impiegare il pacchetto DAAG.
[4] Ovviamente potete procedere con ulteriori approfondimenti impiegando la documentazione del pacchetto [1], ma potete anche valutare al bisogno l'opportunità di ricorrere a qualcuno dei corsi sull'argomento disponibili sul web, cito per tutti un esempio, "Introduction to Data Visualization with ggplot2":
https://www.datacamp.com/courses/introduction-to-data-visualization-with-ggplot2




Nessun commento:
Posta un commento