Per la rappresentazione grafica della distribuzione di una variabile, in alternativa ai kernel density plot possiamo impiegare i più tradizionali istogrammi.
È arcinoto che gli istogrammi sono realizzati raggruppando i valori espressi in una scala numerica continua, riportati in ascisse, in un certo numero di sottoinsiemi (classi) e riportando in ordinate la frequenza dei valori (espressa come numero dei casi, come percentuale dei casi o come probabilità) che cadono in ciascuna classe.
Forse meno noto è che trattandosi della rappresentazione di dati espressi in una scala numerica continua, negli istogrammi le barre che rappresentano le classi devono essere unite tra loro. Questo aspetto li differenzia dai grafici a barre, che sono impiegati per rappresentare dati espressi in forma di scale nominali, scale ordinali e scale numeriche discrete, che non sono scale continue e per questa ragione devono impiegare barre staccate [1].
Vediamo ora una serie di esempi di istogrammi, interessanti in quanto realizzati senza la necessità di installare pacchetti aggiuntivi ma impiegando solamente le funzioni statistiche e grafiche di base di R.
Tutti gli istogrammi mostrano la distribuzione della concentrazione degli eritrociti (globuli rossi) nel sangue rilevata in 202 atleti australiani riportata nella colonna/variabile rcc della tabella ais inclusa nel pacchetto DAAG. Potete scaricare il pacchetto dal CRAN oppure acquisire la tabella seguendo le indicazioni alternative riportate in [2].
Gli esempi sono stati riportati in script separati, in modo che quelli che più interessano possano essere salvati indipendentemente l'uno dall'altro ed essere successivamente ripresi per analizzare i propri dati semplicemente eliminando la prima riga di codice, sostituendo ais$rcc con il nome della propria variabile e adattando opportunamente titoli ed etichette.
Copiate e incollate il primo script nella Console di R e premete ↵ Invio:
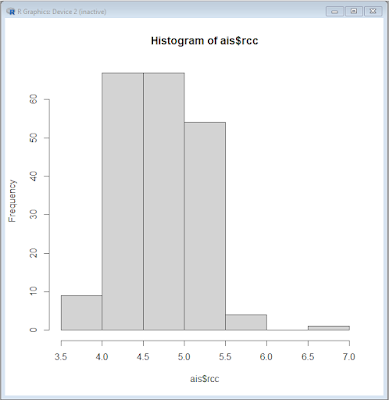
# ISTOGRAMMA SEMPLICE
#
library(DAAG) # carica il pacchetto DAAG che include il set di dati ais
windows() # apre e inizializza una nuova finestra grafica
#
hist(ais$rcc) # traccia l'istogramma
#
Questo è il codice più conciso con cui è possibile realizzare un istogramma con R: prevede di impiegare nella funzione hist(), come unico argomento, l'indicazione dei dati da rappresentare, nel nostro caso ais$rcc ovvero i valori della colonna/variabile rcc della tabella ais. Si tratta di una soluzione minima, che impiega i valori di default previsti per la funzione, e che può essere utile quando si voglia effettuare senza fatica una rapida analisi preliminare dei dati.
Nota bene: nella funzione hist() il valore di default per l'argomento che calcola il numero di classi è breaks="Sturges" che qui porta alla suddivisione dei dati in sole 7 classi. Nei prossimi script vediamo come sia possibile in alternativa:
→ specificare breaks="Scott" oppure breaks="FD" (scelte consigliate);
→ calcolare il numero di classi con sqrt(length(nomedellavariabile)) cioè come radice quadrata sqrt() del numero di dati length(nomedellavariabile), soluzione meno sofisticata delle precedenti ma tendenzialmente adeguata;
→ imporre come numero di classi un valore numerico arbitrario (scelta sconsigliata a meno di avere preventivamente stabilito per altra via il numero opportuno delle classi).
Ora copiate e incollate questo secondo script nella Console di R e premete ↵ Invio:
# ISTOGRAMMA PERSONALIZZATO
#
library(DAAG) # carica il pacchetto DAAG che include il set di dati ais
windows() # apre e inizializza una nuova finestra grafica
#
istog <- hist(ais$rcc, breaks=15, xlim=c(3,7), ylim=c(0,40), col="chartreuse", border=FALSE, main="Istogramma personalizzato", xlab="Eritrociti in 10^12/L", ylab="Frequenza (numero dei casi)") # traccia un istogramma personalizzato
text(cex=0.7, istog$mids, istog$counts, labels=istog$counts, adj=c(0.5, -0.5)) # aggiunge il numero dei casi per ciascuna classe
#
Se nella Console di R digitate help(hist) trovate l'elenco completo degli argomenti che potete impiegare per personalizzare un istogramma e dei quali qui vediamo quelli essenziali:
→ breaks=15 per fissare a 15 il numero delle classi (vedere nota allo script precedente);→ xlim=c(3,7) per fissare limite inferiore e limite superiore dell'asse delle ascisse;
→ ylim=c(0,40) per fissare limite inferiore e limite superiore dell'asse delle ordinate;
→ col="chartreuse" per colorare l'istogramma con uno dei moltissimi colori della tavolozza di R [3];
→ border=FALSE per eliminare il bordo che delimita ciascuna classe;
→ main="..." per riportare il titolo del grafico;
→ xlab="..." e ylab="..." per riportare le legende degli assi.
L'istogramma viene sia tracciato sia salvato nell'oggetto istog. Se nella Console di R digitate
str(istog)
potete visualizzare la struttura dell'oggetto istog
> str(istog)
List of 6
$ breaks : num [1:16] 3.8 4 4.2 4.4 4.6 4.8 5 5.2 5.4 5.6 ...
$ counts : int [1:15] 9 22 26 30 18 38 32 18 5 2 ...
$ density : num [1:15] 0.223 0.545 0.644 0.743 0.446 ...
$ mids : num [1:15] 3.9 4.1 4.3 4.5 4.7 4.9 5.1 5.3 5.5 5.7 ...
$ xname : chr "ais$rcc"
$ equidist: logi TRUE
- attr(*, "class")= chr "histogram"
che contiene le informazioni che ci servono per poter riportare in testa a ciascuna classe il numero di dati in essa contenuti impiegando la funzione text() dell'ultima riga di codice. Nella Console di R digitate help(text) per una spiegazione completa degli argomenti impiegati per ottenere questo risultato.
Nota bene: rispetto all'istogramma precedente, che lasciando il valore di default per l'argomento breaks era suddiviso in 7 classi e mostrava una distribuzione unimodale, la suddivisione dell'istogramma in 15 classi mostra ora una distribuzione bimodale, che tipicamente si osserva quando una variabile/serie di misure è in realtà composta da due sottoinsiemi aventi mode/medie differenti.
Se volete sovrapporre a un istogramma la curva gaussiana corrispondente copiate e incollate nella Console di R questo terzo script e premete ↵ Invio:
# ISTOGRAMMA CON LA CURVA GAUSSIANA CORRISPONDENTE e ordinate in comune
#
library(DAAG) # carica il pacchetto DAAG che include il set di dati ais
windows() # apre e inizializza una nuova finestra grafica
#
hist(ais$rcc, xlim=c(3,7), ylim=c(0,1), freq=FALSE, breaks="FD", col="chartreuse", main="Istogramma con curva gaussiana", xlab="Eritrociti in 10^12/L", ylab="Densità di probabilità") # traccia l'istogramma
x <- seq(min(ais$rcc), max(ais$rcc), length.out=100) # calcola ascisse della curva gaussiana
y <- dnorm(x, mean=mean(ais$rcc), sd=sd(ais$rcc)) # calcola ordinate della curva gaussiana
lines(x, y, col="blue", lwd=1) # sovrappone all'istogramma la curva gaussiana
#
→ freq=FALSE che consente di riportare sull'asse delle ordinate, invece del numero di dati come è accaduto nei due script precedenti, la densità di probabilità, per poter successivamente sovrapporre all'istogramma la curva gaussiana le cui ordinate sono espresse nella stessa scala;
→ breaks="FD" con il quale il numero delle classi viene calcolato impiegando la regola di Freedman-Diaconis [4], che consente di minimizzare la differenza tra l'area dell'istogramma e l'area della gaussiana corrispondente e porta alla suddivisione dell'istogramma in 15 classi.
La curva gaussiana teorica corrispondente ai dati osservati viene calcolata in tre passaggi:
→ con la funzione seq() sono calcolati 100 (length.out=100) valori x delle ascisse compresi tra il valore minimo min(ais$rcc) e il valore massimo max(ais$rcc) della variabile rappresentata - il numero di valori calcolati è stato scelto in base alla risoluzione grafica ma può ovviamente essere aumentato al bisogno;
→ con la funzione dnorm() sono calcolati i valori in ordinate y corrispondenti ai 40 valori x precedenti e aventi la media mean=mean(ais$rcc) e la deviazione standard sd=sd(ais$rcc) della variabile analizzata;
→ con la funzione lines() viene sovrapposta all'istogramma la curva gaussiana passante per i 100 punti di coordinate x,y così calcolati.
Nota bene: risulta evidente come la curva gaussiana, per definizione unimodale, mal si adatta alla distribuzione bimodale dei dati osservabile nell'istogramma.
Ora copiate e incollate nella Console di R questo quarto script e premete ↵ Invio:
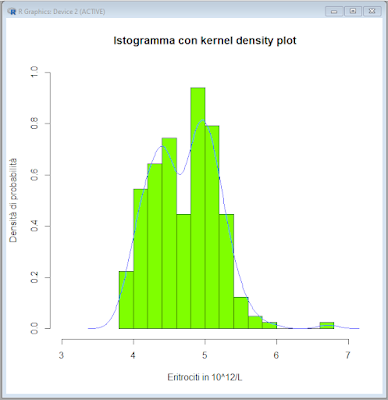
# ISTOGRAMMA CON IL KERNEL DENSITY PLOT CORRISPONDENTE e ordinate separate
#
library(DAAG) # carica il pacchetto DAAG che include il set di dati ais
windows() # apre e inizializza una nuova finestra grafica
#
par(mar=c(5.1,4.1,4.1,5.1)) # aumenta il margine destro da 2.1 a 5.1
hist(ais$rcc, xlim=c(3,7), ylim=c(0,40), freq=TRUE, col="chartreuse", breaks="Scott", main="Istogramma con kernel density plot", xlab="Eritrociti in 10^12/L", ylab="Frequenza (numero dei casi)") # traccia l'istogramma
#
par(new=TRUE, ann=FALSE) # consente la sovrapposizione del grafico successivo
plot(density(ais$rcc), yaxt="n", xlim=c(3,7), ylim=c(0,1), col="blue", lwd=1) # sovrappone all'istogramma il kernel density plot
#
axis(4, col.ticks="blue", col.axis="blue") # riporta sulla destra l'asse delle y del kernel density plot
mtext("Stima kernel della densità di probabilità", side=4, line=3, cex=1, col="blue") # aggiunge la legenda all'asse delle y sulla destra
par(mar=c(5.1,4.1,4.1,2.1)) # ripristina i margini di default
#
→ l'asse delle y sulla sinistra riferito all'istogramma e che riporta la frequenza espressa come numero dei casi;
→ l'asse delle y sulla destra riferito al kernel density plot e che riporta la stima kernel della densità di probabilità.
Ecco le principali osservazioni sugli argomenti e sulle funzioni impiegate in questo script:
→ come prima cosa il margine sulla destra del grafico viene aumentato dal valore di default 2.1 a 5.1 per creare lo spazio necessario per la legenda dell'asse delle y del kernel density plot, posizionato sulla destra;
→ nella funzione hist() viene riportato in ordinate il numero dei casi ponendo freq=TRUE;
→ il numero delle classi viene calcolato con l'argomento breaks="Scott" che fornisce un risultato (15 classi) uguale a quello ottenuto con la regola di Freedman-Diaconis;
→ la funzione par(new=TRUE, ann=FALSE) specifica che il grafico successivo verrà sovrapposto (new=TRUE) ma che non devono essere annotati automaticamente titolo e legende (ann=FALSE), perchè questo lo faremo con le righe successive;
→ con la funzione plot() il kernel density plot viene sovrapposto all'istogramma specificando con yaxt="n" che non deve essere riportato l'asse delle y che si sovrapporrebbe a quello dell'istogramma e che invece noi aggiungeremo ora sulla destra;
→ con la funzione axis() riportiamo l'asse delle y del kernel density plot nella posizione 4 cioè sulla destra (nella grafica di base di R i margini sono 1=margine inferiore, 2=margine sinistro, 3=margine superiore, 4=margine destro) e riportiamo in colore "blue" sia le tacche (col.ticks) sia i corrispondenti valori numerici della scala (col.axis);
→ con la funzione mtext() aggiungiamo sulla destra (side=4) la legenda dell'asse delle y del kernel density plot, posizioniamo l'etichetta "Stima kernel della densità di probabilità" sulla line=3 (la linea 0 contiene le tacche, la 1 contiene i valori numerici, la 2 è volutamente lasciata vuota), riportiamo i caratteri nella dimensione standard (cex=1) e in colore blu (col="blue");
→ con la funzione par() ripristiniamo i margini di default, di fatto riportiamo quello di destra (il quarto valore dell'argomento mar) da 5.1 a 2.1.
Se preferite impiegare un'unica scala sia per l'istogramma sia per il kernel density plot, esprimendo per entrambi le ordinate in termini di densità di probabilità, copiate e incollate nella Console di R questo script e premete ↵ Invio:
# ISTOGRAMMA CON IL KERNEL DENSITY PLOT CORRISPONDENTE e ordinate in comune
#
library(DAAG) # carica il pacchetto DAAG che include il set di dati ais
windows() # apre e inizializza una nuova finestra grafica
#
hist(ais$rcc, xlim=c(3,7), ylim=c(0,1), freq=FALSE, breaks="FD", col="chartreuse", main="Istogramma con kernel density plot", xlab="Eritrociti in 10^12/L", ylab="Densità di probabilità") # traccia l'istogramma
#
par(new=TRUE, ann=FALSE) # consente la sovrapposizione del grafico successivo
plot(density(ais$rcc), xlim=c(3,7), ylim=c(0,1), col="blue", lwd=1) # sovrappone all'istogramma il kernel density plot
#
il codice risulterà semplificato in quanto sono sufficienti tre sole righe con:
→ la funzione hist() per tracciare l'istogramma;
→ la funzione par() per consentire la sovrapposizione del grafico successivo;
→ la funzione plot() per tracciare il kernel density plot.
Se vi accontentate di un'estetica un poco più spartana potete anche ridurre il codice ai minimi termini, copiate e incollate nella Console di R questo script e premete ↵ Invio:
# ISTOGRAMMA CON IL KERNEL DENSITY PLOT CORRISPONDENTE e ordinate in comune
#
library(DAAG) # carica il pacchetto DAAG che include il set di dati ais
windows() # apre e inizializza una nuova finestra grafica
#
hist(ais$rcc, xlim=c(3,7), ylim=c(0,1), freq=FALSE, breaks="FD", col="chartreuse", main="Istogramma con kernel density plot", xlab="Eritrociti in 10^12/L", ylab="Densità di probabilità") # traccia l'istogramma
#
lines(density(ais$rcc), xlim=c(3,7), ylim=c(0,1), col="blue", lwd=1) # sovrappone all'istogramma il kernel density plot
#
Ora sono sufficienti due sole righe di codice con:
→ la funzione hist() per tracciare l'istogramma;
→ la funzione lines() per sovrapporre il kernel density plot.
Nota bene: diversamente dalla distribuzione gaussiana, il kernel density plot, proprio per il modo in cui viene generato [6], descrive bene la distribuzione bimodale dei dati osservabile nell'istogramma.
Per affiancare due istogrammi è sufficiente disporli in due colonne affiancate, copiate e incollate questo script nella Console di R e premete ↵ Invio:
# DUE ISTOGRAMMI AFFIANCATI
#
library(DAAG) # carica il pacchetto DAAG che include il set di dati ais
windows() # apre e inizializza una nuova finestra grafica
#
f <-as.matrix(subset(ais, sex=="f", select=c(rcc))) # estrae il sottoinsieme dei dati con sesso=f
m <-as.matrix(subset(ais, sex=="m", select=c(rcc))) # estrae il sottoinsieme dei dati con sesso=m
par(mfrow=c(1,2)) # grafici organizzati in una riga e due colonne
#
hist(f, xlim=c(3,7), ylim=c(0,30), breaks=sqrt(length(f)), col=rgb(1,0,0,0.5), main="Istogrammi affiancati", xlab="Eritrociti in 10^12/L", ylab="Frequenza (numero dei casi)") # istogramma di f
legend("topright", legend=c("f"), col=c(rgb(1,0,0,0.5)), pt.cex=2, pch=15) # riporta la legenda
hist(m, xlim=c(3,7), ylim=c(0,30), breaks=sqrt(length(m)), col=rgb(0,1,0,0.5), main="", xlab="Eritrociti in 10^12/L", ylab="Frequenza (numero dei casi)") # istogramma di m
legend("topright", legend=c("m"), col=c(rgb(0,1,0,0.5)), pt.cex=2, pch=15) # riporta la legenda
#
Gli istogrammi sono generati con la funzione hist() i cui argomenti di base sono già noti, mentre qui si può notare in aggiunta che:
→ gli istogrammi sono disposti con par(mfrow=c(1,2) in una riga e due colonne;
→ il numero delle classi breaks=sqrt(length(f)) è calcolato come radice quadrata del numero dei dati, il metodo di calcolo tradizionale e più semplice;
→ gli istogrammi prevedono l'impiego di un colore specificato con la funzione rgb(1,0,0,0.5) nella quale il primo numero indica il rosso (red), il secondo il verde (green), il terzo il blu (blu) e il quarto il grado di trasparenza del colore impiegato in una scala che va da 0 a 1;
→ la legenda è posizionata in alto a destra ("topright") e il colore del relativo istogramma viene riportato in un quadrato (pch=15) le cui dimensioni relative sono specificate come pt.cex=2.
Nota bene: se osserviamo attentamente il lato sinistro dell'istogramma della distribuzione dei globuli rossi nel sesso femminile, istogramma che è chiaramente unimodale, e il lato destro dell'istogramma nel sesso maschile, anch'esso unimodale, possiamo riconoscere il lato sinistro e il lato destro dell'istogramma che mostra la distribuzione bimodale dei dati globali negli script precedenti.
Nota bene: non vi piace l'estetica dei due istogrammi così affiancati? Potete "afferrare" con il mouse il lato sinistro della finestra grafica e, tenendo premuto il tasto sinistro del mouse, potete allargarla a piacere!
Ora il salto di qualità nella rappresentazione consiste nel sovrapporre i due precedenti istogrammi unimodali dei dati separati per sesso tracciando due istogrammi sovrapposti e visibili nella loro interezza, senza che uno nasconda l'altro.
Copiate e incollate questo script nella Console di R e premete ↵ Invio:
# DUE ISTOGRAMMI SOVRAPPOSTI
#
library(DAAG) # carica il pacchetto DAAG che include il set di dati ais
windows() # apre e inizializza una nuova finestra grafica
#
f <-as.matrix(subset(ais, sex=="f", select=c(rcc))) # estrae il sottoinsieme dei dati con sesso=f
m <-as.matrix(subset(ais, sex=="m", select=c(rcc))) # estrae il sottoinsieme dei dati con sesso=m
#
hist(f, xlim=c(3,7), ylim=c(0,30), breaks=sqrt(length(f)), col=rgb(1,0,0,0.5), main="Istogrammi sovrapposti", xlab="Eritrociti in 10^12/L", ylab="Frequenza (numero dei casi)") # istogramma di f
hist(m, add=TRUE, xlim=c(3,7), ylim=c(0,30), breaks=sqrt(length(m)), col=rgb(0,1,0,0.5), ) # istogramma di m
legend("topright", legend=c("f","m"), col=c(rgb(1,0,0,0.5), rgb(0,1,0,0.5)), pt.cex=2, pch=15 ) # riporta la legenda
#
Qui l'unica novità è l'argomento add=TRUE che consente di sovrapporre il secondo istogramma al primo, facendo si che entrambi gli istogrammi siano visibili nella loro interezza grazie alla trasparenza del colore assicurata con il quarto valore della funzione rgb() che viene posto uguale a 0.5.
Potete anche considerare per la sovrapposizione di due istogrammi questa estetica alternativa, copiate e incollate questo script nella Console di R e premete ↵ Invio:
# DUE ISTOGRAMMI SOVRAPPOSTI
#
library(DAAG) # carica il pacchetto DAAG che include il set di dati ais
windows() # apre e inizializza una nuova finestra grafica
#
f <-as.matrix(subset(ais, sex=="f", select=c(rcc))) # estrae il sottoinsieme dei dati con sesso=f
m <-as.matrix(subset(ais, sex=="m", select=c(rcc))) # estrae il sottoinsieme dei dati con sesso=m
#
hist(f, xlim=c(3,7), ylim=c(0,30), breaks=sqrt(length(f)), border="red", col="red", density=20, angle=45, main="Istogrammi sovrapposti", xlab="Eritrociti in 10^12/L", ylab="Frequenza (numero dei casi)") # istogramma di f
hist(m, add=TRUE, xlim=c(3,7), ylim=c(0,30), breaks=sqrt(length(m)), border="green4", col="green4", density=20, angle=-45) # istogramma di m
legend("topright", legend=c("f","m"), fill=c("red","green4"), border=c("red","green4"), col=c("red","green4"), density=c(20,20), angle=c(45,-45)) # riporta la legenda
#
Gli argomenti aggiuntivi che è necessario impiegare nella funzione hist() per realizzare questo tipo di trasparenza sono:
→ border=... per specificare il colore del bordo che delimita le barre del primo c("red",...) e del secondo c(...,"green4") istogramma;
→ col=... per il colore da applicare al tratteggio definito con i due argomenti che seguono;
→ density=... per la densità del tratteggio interno alle barre dei due istogrammi;
→ angle=... per l'angolo di pendenza del tratteggio che con un valore positivo dell'angolo (45) nel primo istogramma sarà inclinato da sinistra a destra dal basso in alto, e con un valore negativo dell'angolo (-45) nel secondo istogramma sarà inclinato da sinistra a destra dall'alto in basso.
Nella funzione legend() l'argomento fill=c("red","green4") genera i due riquadri che, impiegando questi stessi quattro argomenti, richiamano colori e aspetto delle barre dei due istogrammi.
Nota bene: dalla sovrapposizione dei due istogrammi unimodali dei dati separati per sesso riotteniamo l'istogramma bimodale della concentrazione degli eritrociti nei dati originari, come volevasi dimostrare.
Potete anche tracciare due istogrammi contrapposti, copiate e incollate quest'ultimo script nella Console di R e premete ↵ Invio:
# DUE ISTOGRAMMI CONTRAPPOSTI
#
library(DAAG) # carica il pacchetto DAAG che include il set di dati ais
windows() # apre e inizializza una nuova finestra grafica
#
f <-as.matrix(subset(ais, sex=="f", select=c(rcc))) # estrae il sottoinsieme di ais con sesso=f
m <-as.matrix(subset(ais, sex=="m", select=c(rcc))) # estrae il sottoinsieme di ais con sesso=m
par(mfrow=c(2,1)) # grafici organizzati in due righe e una colonna
#
par(mar=c(0,5,3,3)) # adatta i margini al grafico superiore
hist(f, xaxt="n", xlim=c(3,7), ylim=c(0,30), breaks=sqrt(length(f)), col=rgb(1,0,0,0.5), las=1, main="Istogrammi contrapposti", xlab="", ylab="Frequenza (numero dei casi)") # istogramma di f
legend("topright", legend=c("f"), col=c(rgb(1,0,0,0.5)), pt.cex=2, pch=15) # riporta la legenda
par(mar=c(5,5,0,3)) # adatta i margini al grafico inferiore
hist(m, xlim=c(3,7), ylim=c(30,0), breaks=sqrt(length(m)), col=rgb(0,1,0,0.5), las=1, main="", xlab="rcc", ylab="Frequenza (numero dei casi)") # istogramma di m
legend("right", legend=c("m"), col=c(rgb(0,1,0,0.5)), pt.cex=2, pch=15) # riporta la legenda
par(mar=c(5.1,4.1,4.1,2.1)) # ripristina i margini di default
#
Qui non c'è molto da aggiungere, se non che si tratta di una estetica alternativa a quella dello script precedente, che può piacere o non piacere e che viene realizzata con alcuni piccoli accorgimenti, come:
→ par(mfrow=c(2,1)) per disporre i grafici in due righe per una colonna quindi in definitiva uno sopra l'altro;
→ par(mar=c(0,5,3,3)) per eliminare il margine inferiore al grafico superiore;
→ xaxt="n" per togliere l'asse delle ascisse al grafico superiore;
→ par(mar=c(5,5,0,3)) per eliminare il margine superiore al grafico superiore;
→ par(mfrow=c(2,1)) per disporre i grafici in due righe per una colonna quindi in definitiva uno sopra l'altro;
→ par(mar=c(0,5,3,3)) per eliminare il margine inferiore al grafico superiore;
→ xaxt="n" per togliere l'asse delle ascisse al grafico superiore;
→ par(mar=c(5,5,0,3)) per eliminare il margine superiore al grafico superiore;
→ ylim=c(30,0) necessario per invertire nel grafico inferiore l'asse delle y rispetto al grafico superiore, nel quale abbiamo il più usuale ylim=c(0,30).
Infine ricordo che nel post Salvare i grafici di R in un file è riportato uno script che consente di salvare i grafici sotto forma di file .bmp, .jpeg, .png, .pdf, .ps per poterli stampare, archiviare, inserire in una pubblicazione, in un post o in un sito web.
Conclusione: gli istogrammi - un rappresentazione grafica semplice e tradizionale della distribuzione di una variabile espressa in una scala numerica continua - anche se possono sembrare uno strumento vetusto, quando impiegati con una opportuna suddivisione in classi, adeguatamente rappresentati e attentamente esaminati, offrono un importante ausilio nell'analisi dei dati.
----------
[2] Vedere nel post Il set di dati ais come acquisire i dati della tabella ais anche senza istallare il pacchetto DAAG. La concentrazione degli eritrociti è espressa in 1012/L o in 106/µL, le due unità di misura impiegate nella pratica medica, che sono numericamente identiche.
[4] Vedere: Freedman–Diaconis rule.
https://en.wikipedia.org/wiki/Freedman%E2%80%93Diaconis_rule
[5] Vedere anche il post Adattare i margini a un grafico.
[6] Vedere il post Kernel density plot.








Nessun commento:
Posta un commento