La regressione lineare semplice (regressione bivariata) costruita sul piano cartesiano
y = a + b·x
può essere estesa a uno spazio n-dimensionale: abbiamo così la regressione lineare multipla nella quale la variabile dipendente y viene fatta dipendere non più da una, bensì da k variabili indipendenti, indicate come x₁, x₂, x₃ … xₖ secondo l'equazione
y = a + b₁·x₁ + b₂·x₂ + b₃·x₃ + ... + bₖ·xₖ
Il risultato è rappresentato quindi da una intercetta a e da tanti coefficienti angolari b (indicati con b₁, b₂, b₃ ... bₖ) quante sono le variabili indipendenti (x₁, x₂, x₃ ... xₖ) che contribuiscono a determinare il valore della variabile dipendente y.
Come esempio impieghiamo il set di dati ais nel quale, tra i dati ematologici rilevati in un gruppo di atleti australiani, abbiamo tre grandezze:
→ la concentrazione degli eritrociti (globuli rossi) espressa in milioni per microlitro di sangue (10⁶/µL) e contenuta nella variabile rcc;
→ il valore ematòcrito cioè la frazione del volume del sangue occupata dagli eritrociti espressa in percentuale (%) e contenuta nella variabile hc;
→ la concentrazione dell'emoglobina espressa in grammi per decilitro di sangue (g/dL) e contenuta nella variabile hg.
Le tre grandezze sono strettamente legate tra loro dal punto di vista biologico, in quanto gli eritrociti hanno ciascuno un certo volume e contengono ciascuno una data quantità di emoglobina: pertanto all'aumentare del numero di eritrociti aumenta la frazione percentuale del volume del sangue occupata dagli eritrociti (il valore ematòcrito) e aumenta la quantità di emoglobina. Mentre la diminuzione dell'ematòcrito e la diminuzione dell'emoglobina sono indice della diminuzione del numero degli eritrociti.
L'esempio è stato scomposto in una serie di script indipendenti - onde facilitare il riutilizzo del codice per analizzare i propri dati - che impiegano i pacchetti aggiuntivi gvlma, car e rgl, che devono essere scaricati dal CRAN. Accertatevi di avere installato inoltre il pacchetto DAAG che contiene i dati impiegati nello script, o in alternativa procedete come indicato nel post Il set di dati ais.
Iniziamo con una analisi esplorativa preliminare dei dati, copiate questo primo script, incollatelo nella Console di R e premete ↵ Invio:
# REGRESSIONE LINEARE MULTIPLA - analisi esplorativa preliminare dei dati
#
library(DAAG) # carica il pacchetto che include il set di dati ais
library(car) # carica il pacchetto per la grafica
mydata <- ais[,c(1,3,4)] # le colonne 1, 3, 4 contengono le variabili rcc, hc, hg
windows() # apre e inizializza una nuova finestra grafica
#
scatterplotMatrix(~rcc+hc+hg, col="black", pch=20, regLine=list(method=lm, lty=1, lwd=2, col="chartreuse3"), smooth=FALSE, diagonal=list(method="histogram", breaks="FD"), main="Istogrammi e grafici xy con rette di regressione", data=mydata) # istogrammi e grafici xy con rette di regressione
#
Dopo avere caricato i pacchetti necessari, copiato nell'oggetto mydata le tre variabili che ci interessano, aperto e inizializzato una nuova finestra grafica, l'analisi preliminare dei dati viene effettuata con la funzione scatterplotMatrix() nella quale:
→ ~rcc+hc+hg specifica le variabili da rappresentare, che sono la concentrazione degli eritrociti rcc, il valore ematòcrito hc e la concentrazone di emoglobia hg;
→ col="..." specifica il colore dei grafici;
→ pch=... specifica il tipo di simbolo da impiegare (un piccolo cerchio pieno);
→ per la retta di regressione regLine sono impiegati il modello lineare (lm), una linea continua (lty=1) con uno spessore doppio (lwd=2) e un colore (col="chartreuse3"), mentre l'argomento smooth=FALSE fa si che non venga aggiunta ai grafici la rappresentazione della funzione non lineare prevista di default nel pacchetto car;
→ l'argomento diagonal=list() indica la rappresentazione della variabile riportata nella diagonale, che qui è l'istogramma (method="histogram") con il numero di classi (breaks="FD") calcolato mediante la regola di Freedman-Diaconis.
Da notare che le espressioni che è possibile impiegare per l'argomento diagonal=... sono:
→ list(method="boxplot");
→ list(method="density", bw="nrd0", adjust=1, kernel="gaussian", na.rm=TRUE);
→ list(method="histogram");
→ list(method="oned");
→ list(method="qqplot").
Vista la relazione lineare molto buona che intercorre tra le variabili sembra ragionevole continuare ed effettuare il calcolo della regressione lineare multipla, copiate questo secondo script, incollatelo nella Console di R e premete ↵ Invio:
# REGRESSIONE LINEARE MULTIPLA - calcolo della regressione lineare multipla
#
library(DAAG) # carica il pacchetto che include il set di dati ais
mydata <- ais[,c(1,3,4)] # le colonne 1, 3, 4 contengono le variabili rcc, hc, hg
reglin <- lm(rcc~hg+hc, data=mydata) # calcolo della regressione
#
coefficients(reglin) # intercetta e coefficienti angolari
confint(reglin, level=0.95) # intervalli di confidenza al 95%
#
La funzione lm(...) viene impiegata per calcolare la regressione lineare multipla specificando con l'espressione rcc~hg+hc la variabile dipendente (rcc), le due variabili indipendenti (hg e hc) e l'equazione
rcc = a + b₁·hg + b₂·hc
quindi possiamo ricavare con la funzione coefficients() i valori dell'intercetta (a), del coefficiente angolare di hg (b₁) e del coefficiente angolare di hc (b₂):
(Intercept) hg hc
-0.24269563 0.03283747 0.10403395
e riportare l'equazione della regressione lineare multipla come
rcc = -0.24269563 + 0.03283747·hg + 0.10403395·hc
Successivamente con la funzione confint() sono riportati gli intervalli di confidenza al 95% dell'intercetta e dei due coefficienti angolari:
2.5 % 97.5 %
(Intercept) -0.53163317 0.04624192
hg -0.02459817 0.09027311
hc 0.08267073 0.12539718
Da notare che gli intervalli di confidenza forniscono un test di significatività per intercetta e coefficienti angolari:
→ dato un intervallo di confidenza al 95% che va da -0.53163317 a 0.04624192 (che quindi include il valore 0) l'intercetta non è significativamente diversa da 0, cosa molto logica in quanto al diminuire fino ad azzerarsi dei valori di emoglobina ed ematòcrito deve necessariamente diminuire e azzerarsi il numero degli eritrociti;
→ con un intervallo di confidenza al 95% che va da -0.02459817 a 0.09027311 (che quindi include il valore 0) il coefficiente angolare della concentrazione di emoglobina hg non è significativamente diversa da 0, cosa meno logica in quanto non solo dal punto di vista biologico deve esistere una proporzionalità tra numero degli eritrociti e concentrazione dell'emoglobina, ma anche nell'analisi esplorativa preliminare dei dati riportata con lo script precedente risulta evidente la proporzionalità tra numero degli eritrociti e concentrazione dell'emoglobina che è una dato di fatto dal punto di vista biologico;
→ con un intervallo di confidenza al 95% che va da 0.08267073 a 0.12539718 (che quindi non include il valore 0) il coefficiente angolare del valore ematòcrito hc risulta significativamente diverso da 0.
Come detto all'inizio, oltre ad effettuare una analisi esplorativa preliminare dei dati è opportuno sottoporre i risultati ad una valutazione critica a posteriori, che effettuiamo ora sotto forma di una diagnostica numerica della regressione lineare multipla, copiate il terzo script, incollatelo nella Console di R e premete ↵ Invio:
# REGRESSIONE LINEARE MULTIPLA - diagnostica numerica della regressione
#
library(gvlma) # carica il pacchetto per la diagnostica numerica
library(car) # carica il pacchetto per la grafica
library(DAAG) # carica il pacchetto che include il set di dati ais
mydata <- ais[,c(1,3,4)] # le colonne 1, 3, 4 contengono le variabili rcc, hc, hg
reglin <- lm(rcc~hg+hc, data=mydata) # calcolo della retta di regressione lineare multipla
#
summary(gvlma(reglin)) # test globale per l'assunto di linearità
outlierTest(reglin) # valore p di Bonferroni per la presenza di dati aberranti (outliers)
#
Ecco la prima parte del riepilogo dei risultati fornito dalla funzione summary():
Call:
lm(formula = rcc ~ hg + hc, data = mydata)
Residuals:
Min 1Q Median 3Q Max
-0.55791 -0.11284 -0.00699 0.09950 0.53595
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) -0.24270 0.14652 -1.656 0.0992 .
hg 0.03284 0.02913 1.127 0.2609
hc 0.10403 0.01083 9.603 <2e-16 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 0.1744 on 199 degrees of freedom
Multiple R-squared: 0.8565, Adjusted R-squared: 0.855
F-statistic: 593.8 on 2 and 199 DF, p-value: < 2.2e-16
Il valore di probabilità Pr(>|t|) ottenuto con il test t di Student conferma (ovviamente) i risultati della significatività ricavati dagli intervalli di confidenza riportati sopra, per i quali valgono le considerazioni già fatte. E se la significatività statistica non è coerente con una realtà biologica assodata, dovrà ovviamente essere quest'ultima a prevalere: per cui assumiamo che l'equazione calcolata sia, fino a prova contraria, una descrizione sufficientemente adeguata della relazione di funzione che intercorre tra le tre variabili esaminate.
Che questo sia vero ce lo conferma la funzione gvlma(), acronimo di global validation of linear models assumptions, che fornisce questi risultati:
→ i dati sono distribuiti in modo lineare (Global Stat), assunto accettabile;
→ i dati non presentano asimmetria (Skewness), assunto accettabile;
→ i dati non presentano curtosi (Skewness), assunto accettabile;
→ nell'ambito dei valori osservati la varianza è costante (Heteroscedasticity), assunto accettabile.
ASSESSMENT OF THE LINEAR MODEL ASSUMPTIONS
USING THE GLOBAL TEST ON 4 DEGREES-OF-FREEDOM:
Level of Significance = 0.05
Call:
gvlma(x = reglin)
Value p-value Decision
Global Stat 6.5801 0.15981 Assumptions acceptable.
Skewness 0.5149 0.47304 Assumptions acceptable.
Kurtosis 0.7559 0.38461 Assumptions acceptable.
Link Function 3.9844 0.04592 Assumptions NOT satisfied!
Heteroscedasticity 1.3249 0.24972 Assumptions acceptable.
L'assenza nei dati di asimmetria e di curtosi, e la loro omoschedasticità, sono condizioni sufficienti per garantire la loro adeguata descrizione mediante una regressione lineare. Il primo test (Global Stat) di valutazione globale degli assunti di linearità, che sostanzialmente comprende tutti e tre i test precedenti in forma compatta, li conferma. In questo contesto il test rimanente e l'unico discordante (Link Function) può essere ignorato.
Con la funzione outlierTest() viene effettuato il test di Bonferroni, che non evidenzia dati anomali (outliers) ma segnala il dato 78 come quello che si discosta maggiormente dagli altri:
No Studentized residuals with Bonferroni p < 0.05
Largest |rstudent|:
rstudent unadjusted p-value Bonferroni p
78 -3.28855 0.0011917 0.24073
In tema di valutazione critica a posteriori, aggiungiamo ora alla diagnostica numerica anche una diagnostica grafica della regressione lineare multipla, per effettuarla copiate questo quarto script, incollatelo nella Console di R e premete ↵ Invio:
# REGRESSIONE LINEARE MULTIPLA - diagnostica grafica della regressione
#
library(car) # carica il pacchetto per la grafica
library(DAAG) # carica il pacchetto che include il set di dati ais
mydata <- ais[,c(1,3,4)] # le colonne 1, 3, 4 contengono le variabili rcc, hc, hg
reglin <- lm(rcc~hg+hc, data=mydata) # calcolo della retta di regressione lineare multipla
#
windows() # apre e inizializza una nuova finestra grafica
qqPlot(reglin, main="Grafico dei quantili per i residui", xlab="t-quantili", ylab="Residui studentizzati") # grafico globale per linearità e dati aberranti
#
windows() # apre e inizializza una nuova finestra grafica
ceresPlots(reglin, ask=FALSE, main="Grafico di Ceres", ylab="Residui di Ceres (rcc)") # grafico di Ceres per la valutazione separata della linearità di hg e hc vs. rcc
#
windows() # apre e inizializza una nuova finestra grafica
plot(mydata$rcc, reglin$fitted.values, xlim=c(4,7), ylim=c(4,7)) # grafico valori originari vs valori stimati
abline(0, 1, lty=2) # linea di indentità y = x
#
La funzione qqPlot() riporta una distribuzione dei residui lineare, evidenziando nel contempo i dati 78 e 161 come quelli che dei quali sarebbe utile controllare la validità, ovvero che sono situati in intervalli di valori per i quali potrebbe essere opportuno acquisire più dati.
La funzione ceresPlot() genera due grafici di Ceres separati per emoglobina ed ematòcrito, e consente di visualizzare una relazione lineare tra ematòcrito ed eritrociti migliore di quella che intercorre tra emoglobina ed eritrociti, confermando in un certo senso i valori di significatività riportati sopra.
La diagnostica grafica della regressione lineare multipla si conclude con questo grafico minimalista, ma che entra nell'essenza dei risultati.
Il grafico, generato con la funzione plot(), riporta in ascisse i valori originari della concentrazione degli eritrociti (mydata$rcc) e in ordinate i corrispondenti valori reglin$fitted.values ricalcolati da hg e hc impiegando l'equazione della regressione lineare multipla ove
reglin$fitted.values = -0.24269563 + 0.03283747·hg + 0.10403395·hc
La linea di identità y = x che viene riportata evidenzia, senza la necessità di ulteriori statistiche, che l'informazione acquisita misurando la concentrazione dell'emoglobina e l'ematòcrito può essere compressa in un unico valore, che fornisce una stima mediamente adeguata della concentrazione degli eritrociti.
Nota: con "mediamente adeguata" si intende che, data la misura diretta della concentrazione degli eritrociti riportata in ascisse, la sua misura indiretta - ottenuta misurando emoglobina ed ematòcrito ed applicando la regressione lineare multipla - comporta una perdita di informazione [aumento dell'incertezza della misura] che si riflette nella "dispersione media" dei valori sull'asse delle ordinate. Se non ci fosse perdita di informazione i punti sarebbero perfettamente allineati sulla retta y = x.
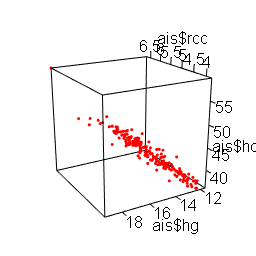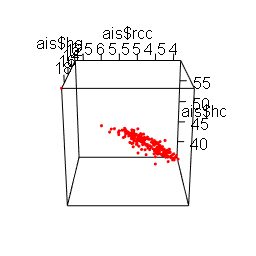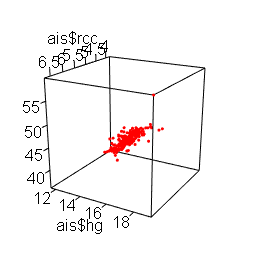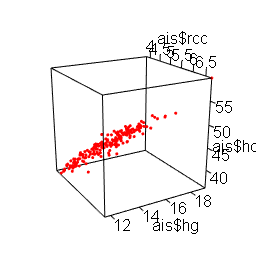
Ora, visto che con tre variabili la cosa è ancora fattibile, non resta che aggiungere un grafico tridimensionale (3D).
Copiate quest'ultimo script, incollatelo nella Console di R e premete ↵ Invio:
# GRAFICO 3D ANIMATO con il pacchetto rgl
#
library(rgl) # carica il pacchetto per la grafica 3D
library(DAAG) # carica il pacchetto che include il set di dati ais
mydata <- ais[,c(1,3,4)] # le colonne 1, 3, 4 contengono le variabili rcc, hc, hg
windows() # apre e inizializza una nuova finestra grafica
#
# grafico 3D animato
#
plot3d(mydata$rcc, y=mydata$hg, z=mydata$hc, type="p", col="red", size=3) # realizza il grafico
play3d(spin3d(axis=c(0,0,1), rpm=10), duration=10) # anima il grafico
#
Dopo avere caricato il pacchetto necessario con il comando library(rgl) il grafico viene realizzato con la funzione plot3d().
Quindi con la funzione play3d() viene realizzata una animazione della durata di 10 secondi (duration=10) con i parametri specificati dalla funzione spin3d() e cioè l'asse sul quale fare ruotare l'animazione, nel nostro caso l'asse delle z (0,0,1), e il numero di rotazioni al minuto (rpm=10).
Quando l'animazione che compare nella finestra "RGL device" aperta nella Console di R dallo script si ferma, se “afferrate” il grafico 3D facendo click con il tasto sinistro del mouse e tenendolo premuto senza rilasciarlo potete ruotarlo a vostro piacimento muovendo il mouse.
Anche se non aggiunge nulla di nuovo, il grafico [1] ci conforta documentando la linearità della relazione tra le tre variabili.
Conclusione: mettere in relazione tra di loro troppe variabili contemporaneamente può portare a risultati di difficile comprensione. Tuttavia una analisi esplorativa preliminare dei dati e una attenta valutazione critica a posteriori dei risultati possono aiutare a identificare i casi nei quali l'impiego della regressione lineare multipla può essere appropriato.
----------
[1] Trovate altre modalità di rappresentazione sotto forma di grafici tridimensionali nel post Grafici 3D.